近年来,RNA-seq 以及 single cell RNA-seq 技术越来越多地被运用于多个研究领域,包括神经研究领域。通常在进行 RNA-seq 获得组织和细胞的高通量转录组数据后,研究人员会通过生物信息学分析,发现并揭示在特定组织细胞类型、生物学过程以及疾病发生发展中的标志性基因和基因表达调控机制。
RNAscope® 技术是一种高灵敏度、高特异性、高稳定性的新型原位检测技术,并可在一张切片上同时检测 1~4 个 RNA,因而可以在检测目标 RNA 的同时,表征特定细胞亚群。其操作流程非常简单,和 IHC/IF 操作类似,仅需 1 天时间即可完成,适用于任意种属、任意组织、任意基因的原位检测,可以高质量地对测序结果进行验证和完善:
用可视化的方式验证目标 RNA 及其基因表达特征
以单细胞水平的分辨率解析基因表达和多基因共表达情况
在组织环境中解析某种细胞类型或状态的空间分布
鉴别适当的单细胞以便进行高质量的转录组分析
本文将举例介绍利用 RNAscope® 技术结合 RNA-seq 技术对中枢神经系统神经细胞分类及验证应用。
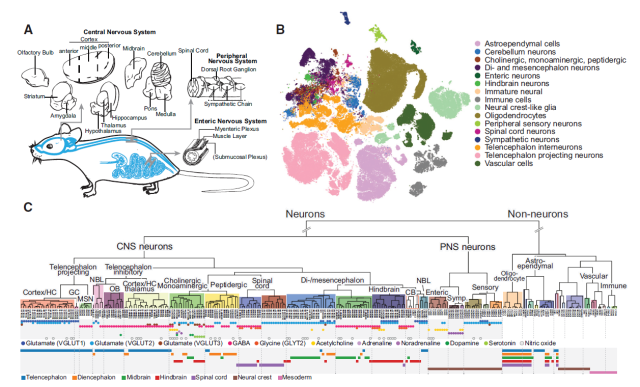
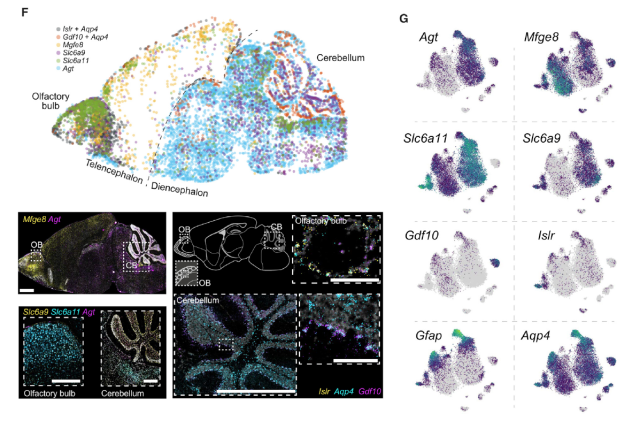
瑞典卡罗林斯卡学院的 Sten Linnarsson 团队 [1],对小鼠中枢神经系统和外周神经系统 19 个部位、共 509,876 个细胞进行单细胞 RNA-seq,从而建立小鼠神经系统细胞图谱。作者将不同类型的细胞对应在空间分布,形成了分层的、数据驱动的神经细胞分类方法。结果发现神经元是最多样化的,可按照发育解剖部位和神经递质、神经肽的表达进行分类。其多样性主要由编码细胞命运、突触连接、神经传递和膜电位的基因驱动的。作者发现了 7 类截然不同的、受区域限制的星形胶质细胞,其分类与关键谷氨酸和甘氨酸的空间分布相关。每一类星形胶质细胞均有自己特异性的基因。作者使用了 RNAscope® 技术进行了原位检测的验证。少突胶质细胞则具有相对较少的多样性。此研究结果为了解哺乳动物神经系统的分子结构奠定了基础,并对特定细胞的遗传操作提供了参考和可能。
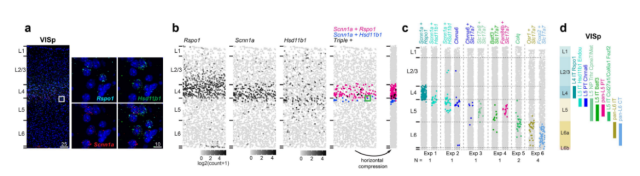
新皮层包含许多细胞类型,它们被分成层和功能不同的区域。来自 Allen Institute for Brain Science 的 Bosiljka Tasic 团队 [2] 为了研究小鼠新皮层细胞类型的多样性,通过对小鼠新皮层的初级视觉皮层和前外侧运动皮层的 23,822 个细胞进行深度的单细胞 RNA-seq,分析确定了 133 种细胞类型。研究发现,几乎所有类型的 GABA(γ-氨基丁酸)神经元在这两个区域共享。而大多数类型的谷氨酸能神经元在这两个区域之一被发现。结合 scRNA-seq 和逆行标记投射研究,作者将不同的谷氨酸能神经元与其长程投射特异性相匹配,并使用了 RNAscope® 技术验证不用的谷氨酸能神经元在皮层里的分布。此研究建立了成年小鼠皮层不同功能区域的皮质细胞类型的联合转录和投影分类,这为描绘大脑神经细胞图谱迈进了重要的一步。
加州大学的 Kung Zhang 团队 [3],从尸检大脑皮层的六个不同区域获得了 3227 组单个神经元转录组数据,并使用迭代聚类和分类的方法,确定了 16 个神经元亚型。作者根据已知的分子标记和皮层结构进行了进一步的注释,并使用 RNAscope® 技术证实了在大脑皮层中特异性的标记物的亚型特异性和分层特异性表达。
约翰霍普金斯医学院的 Maxime Cheve´ e 团队 [4],利用单细胞 RNA-seq 技术,分析躯体感觉皮层 L6 皮质丘脑神经元(L6CThNs)的基因表达谱,发现有两类亚型 L6CThNs,并进一步用 RNAscope® 技术来验证差异候选基因在 Ntsr1-Cre;tdTomato mice 小鼠感觉皮层的 L6CThNs 和非 L6CThNs 中的 mRNA 表达情况。 结果显示 Lamp5, Serpini1, 和 Gabra5 的表达依赖于神经亚型和定位的共同作用。而 Pantr1 的表达在 L6CThNs 及非 L6CThNs 区域出现不同变化。以上结果说明两类 L6CThNs 神经元亚型的基因表达差异与两类 L6CThNs 在 L6 皮层亚定位及大范围轴突投射模式相关的。其各自的转录图谱反映了其轴突投射模式、皮层内位置和神经元的活动状态。
【参考文献】
1. Zeisel, A.H., Hannah; Lönnerberg, Peter; Johnsson, Anna; Memic, Fatima; van der Zwan, Job; Häring, Martin; Braun, Emelie; Borm, Lars E.; La Manno, Gioele; Codeluppi, Simone; Furlan, Alessandro; Lee, Kawai; Skene, Nathan; Harris, Kenneth D.; Hjerling-Leffler, Jens; Arenas, Ernest; Ernfors, Patrik; Marklund, Ulrika; Linnarsson, Sten, Molecular Architecture of the Mouse Nervous System. Cell, 2018. 174(4): p. 999-1014.e22.
2. Tasic, B.Y., Z.; Graybuck, L. T.; Smith, K. A.; Nguyen, T. N.; Bertagnolli, D.; Goldy, J.; Garren, E.; Economo, M. N.; Viswanathan, S.; Penn, O.; Bakken, T.; Menon, V.; Miller, J.; Fong, O.; Hirokawa, K. E.; Lathia, K.; Rimorin, C.; Tieu, M.; Larsen, R.; Casper, T.; Barkan, E.; Kroll, M.; Parry, S.; Shapovalova, N. V.; Hirschstein, D.; Pendergraft, J.; Sullivan, H. A.; Kim, T. K.; Szafer, A.; Dee, N.; Groblewski, P.; Wickersham, I.; Cetin, A.; Harris, J. A.; Levi, B. P.; Sunkin, S. M.; Madisen, L.; Daigle, T. L.; Looger, L.; Bernard, A.; Phillips, J.; Lein, E.; Hawrylycz, M.; Svoboda, K.; Jones, A. R.; Koch, C.; Zeng, H., Shared and distinct transcriptomic cell types across neocortical areas. Nature, 2018. 563(7729): p. 72-78.
3. Lake, B.B.A., R.; Kaeser, G. E.; Salathia, N. S.; Yung, Y. C.; Liu, R.; Wildberg, A.; Gao, D.; Fung, H. L.; Chen, S.; Vijayaraghavan, R.; Wong, J.; Chen, A.; Sheng, X.; Kaper, F.; Shen, R.; Ronaghi, M.; Fan, J. B.; Wang, W.; Chun, J.; Zhang, K., Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science, 2016. 352(6293): p. 1586-90.
4. Chevee, M.R., J. J.; Cannon, G. H.; Brown, S. P.; Goff, L. A., Variation in Activity State, Axonal Projection, and Position Define the Transcriptional Identity of Individual Neocortical Projection Neurons. Cell Rep, 2018. 22(2): p. 441-455.
文章来源:ACD 中国
